Identification of Italian ecotypes of Juglans regia L. by molecular, morphological and biochemical markers
Forest@ - Journal of Silviculture and Forest Ecology, Volume 3, Pages 598-609 (2006)
doi: https://doi.org/10.3832/efor0408-0030598
Published: Dec 18, 2006 - Copyright © 2006 SISEF
Research Articles
Guest Editors: 5° SISEF Congress (Grugliasco, TO - 2005)
« Forests and Society - Changes, Conflicts, Sinergies »
Collection/Special Issue: E. Lingua, R. Marzano, G. Minotta, R. Motta, A. Nosenzo, G. Bovio
Abstract
Juglans regia L. is a multipurpose species important for quality wood and fruit production. In order to contrast the erosion and to properly conserve, manage and revaluate the genetic resources of Italian walnut, possible ecotypes, naturally adapted and still present in Italy have been researched. Leaves and fruits have been sampled in Campania region, localities of Montella, Cervinara, Fisciano, and in Abruzzo region, localities of Sulmona, Pescasseroli, Villetta Barrea, and Civitella Alfedena. The sites are located at different altitudes and climatic conditions. Materials have been collected on a total of 276 plants. Molecular, morphological and preliminary biochemical analyses have been carried out on this germplasm and on material belonging to 80 plants of 4 famous Italian walnut varieties (Bleggiana and Feltrina, North Italy; Sorrento and Malizia, Southern Italy), in order to have a comparison model. 134 ISSR, morphological and biochemical data have shown peculiar characters for Montella and Pescasseroli in comparison with the other accessions. Because of the peculiar environmental conditions of their locations, the effect of the temperature on the fruit development and fatty acid contents, it is possible to suppose that Montella and Pescasseroli are ecotypes which could be utilised as essential fat acid source and as material for afforestation of mountain zones.
Keywords
Juglans regia, Ecotypes, Molecular markers, Morphological markers, Biochemical markers
Introduzione
Juglans regia è una specie con utilizzi molteplici, di notevole importanza sia per la produzione di legno pregiato, sia di frutti. La vasta diffusione della specie è dovuta principalmente ai frequenti scambi di semi, avvenuti nel corso dei secoli, tra Paesi e regioni diverse. Originaria dell’Asia centrale, giunse in Europa probabilmente attraverso le antiche vie della seta che collegavano Cina, India, Persia, Turchia, Grecia ([16]) e paesi occidentali. I romani, che coltivavano il noce fin dal periodo dei Re di Roma (VIII-VII secolo a.C.) lo diffusero in Germania, in Spagna, in Francia e da qui in Inghilterra ([5]). Nonostante tali informazioni testimonino come la nocicoltura in Italia sia un’antica tradizione, ormai da alcuni anni si registra nel nostro Paese una minore produzione di frutto. Ciò può essere imputabile sia alla mancanza di adeguati programmi di miglioramento genetico del materiale italiano, sia alla competitività sul mercato di noci importate da paesi stranieri (soprattutto USA e Cina). Inoltre, per i nuovi impianti da frutto, sono spesso oggi utilizzate varietà straniere, specialmente francesi e californiane, soprattutto perché particolarmente adatte alla raccolta meccanica. Gli impianti, se effettuati con una singola cultivar, sono più soggetti a malattie molto dannose per la produttività e per le piante stesse. Per rilanciare la coltivazione del noce italiano sarebbe perciò di grande aiuto migliorare il germoplasma nazionale, magari riscoprendo e valorizzando quegli ecotipi e/o varietà locali che, a causa della scarsa resa, la notevole grandezza delle piante, nonché l’elevato costo della manodopera necessaria alla raccolta del frutto, sono abbandonate o neglette. Tale abbandono è tra le principali cause del depauperamento delle risorse genetiche e con esso della perdita di geni potenzialmente utili ([14]).
In passato la caratterizzazione di provenienze e varietà si basava sostanzialmente sullo studio di caratteri morfologici, fenologici e fisiologici che sono spesso in stretta relazione con le condizioni pedoclimatiche. Lo sviluppo delle recenti tecniche per l’analisi diretta del DNA ha offerto nuovi mezzi di valutazione. Studi di fingerprinting sul noce sono stati effettuati mediante marcatori isoenzimatici ([4], [2], [38], [14]) e molecolari come RFLP ([12], [13]), RAPD ([26], [30], [1]), ISSR ([34]) e microsatelliti ([33], [15]).
Negli ultimi decenni alcuni studi hanno evidenziato l’importanza del frutto del noce per la salute umana ([40], [43], [41], [6], [3]); in particolare l’acido linoleico (w-6) e l’acido linolenico (w-3), acidi grassi polinsaturi essenziali in esso presenti, sembrano avere un effetto positivo sul sistema cardio-circolatorio poiché contrastano l’aumento di colesterolo nel sangue ([11]).
Lo studio congiunto di caratteri quantitativi (morfologici, adattativi) e qualitativi (marcatori molecolari) è stato spesso considerato e l’eventuale associazione e relazione tra essi è di grande importanza scientifica e pratica.
Nel presente lavoro sono riportati dati preliminari di uno studio condotto con lo lo scopo di identificare e caratterizzare potenziali ecotipi italiani di J. regia e di acquisire informazioni utili per la loro valorizzazione. Per tale ricerca è stato usato un approccio multidisciplinare basato sull’analisi molecolare (ISSR) delle provenienze e la valutazione di alcuni caratteri morfologici e biochimici del frutto. Sono stati usati gli ISSR poiché tali marcatori, che si basano sull’amplificazione casuale di frammenti di DNA genomico mediante primers contenenti ognuno un differente motivo microsatellitare ripetuto ([42]), non richiedono la conoscenza della regione amplificata, permettono un’analisi rapida e relativamente poco costosa; sono polimorfici, riproducibili e sono largamente usati in genetica di popolazione. I caratteri morfologici considerati sono tra quelli comunemente usati per la caratterizzazione varietale, mentre i caratteri biochimici, quali olio, proteine totali e acidi grassi, sono importanti per le qualità nutritive del frutto.
Materiali e Metodi
Campionamento
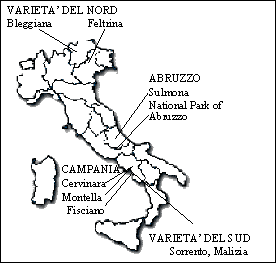
Il campionamento é stato eseguito nelle regioni Campania ed Abruzzo (Fig. 1). La Campania, ove la coltivazione del noce è storicamente provata fin dal 1° secolo d.C. dai resti di Pompei ed Ercolano, è la “patria” della più famosa varietà italiana “Sorrento” e fornisce al mercato circa il 70% del frutto italiano. Anche l’Abruzzo risulta tra le regioni maggiori produttrici di noci ([28]) e include, come la Campania, la J. regia nella lista delle specie indigene o naturalizzate ammesse dal regolamento UE 2080. Campania ed Abruzzo, divise dagli Appennini che impediscono il flusso genico naturale tra le popolazioni, sono classificate come regioni con caratteristiche ecologiche affini ([9]). In ambedue le regioni i campionamenti sono stati eseguiti in aree con elevata concentrazione di piante (Malvolti, osservazione personale) nate, secondo le informazioni del Corpo Forestale e dei proprietari delle fattorie, da seme locale. In 10 differenti siti (Tab. 1), in primavera, sono state collezionate foglie giovani da un totale di 276 piante e, all’inizio dell’autunno, semi sulla metà degli individui. Tale difformitàè principalmente dovuta al fatto che non tutti i proprietari delle piante hanno permesso la raccolta delle noci, così come anche in alcuni siti qualche individuo era stato abbattuto. Le foglie sono state immediatamente immerse in azoto liquido e quindi conservate a -80°C; i frutti sono stati liberati dal mallo e conservati a +4°C.
Tab. 1 - Localizzazione geografica, temperatura media annua, precipitazioni medie annue di 10 siti in Campania e Abruzzo e delle zone d’origine delle quattro varietà-modello. Fonti: (1) www.sito.regione.campania.it: stazione di Baronissi; (2) www.stapacepicaavellino.com: stazione di Mirabella Eclano; (3) www.arssa.abruzzo.it
| Regione | Provincia | Comune | Temp. media annua (°C) | Prec. medie annue (mm) | Localizzazione (lat., long.) | Altitudine (m s.l.m.) |
Accessioni |
|---|---|---|---|---|---|---|---|
| Campania | Salerno | Fisciano1 | 15.8 | 1137 | 40°42’N;14°50’E | 280 | Trescine |
| Avellino | Cervinara2 | 14.8 | 567.3 | 41°03’N;14°40’E | 284 | Mesanole | |
| Montella | 12.8 | 1341.3 | 40°48’N;15°00’E | 670 | Montella | ||
| Abruzzo | Aquila | Sulmona3 (e dintorni) |
13.7 | 615 | 41°08’N;13°51’E | 330 | Pratola Peligna |
| 42°03’N;13°50’E | 430 | Valle Corvo | |||||
| 42°01’N;13°51’E | 500 | Contrada Chiuse | |||||
| 42°12’N;13°45’E | 685 | Navelli | |||||
| Parco Nazionale dell’Abruzzo3 | 7.8 | 1520 | 41°47’N;13°47’E | 1000 | Villetta Barrea | ||
| 41°45’N;14°01’E | 1100 | C.Alfedena | |||||
| 41°49’N;13°46’E | 1200 | Pescasseroli | |||||
| Varietà-modello | Provincia | Comune | Temp. media annua (°C) | Prec. medie annue (mm) | Localizzazione (lat., long.) | Altitudine (ms.l.m.) | |
| Sorrento | Napoli | Sorrento | ca. 16 | 840 mm | 40°55’N;14°48’E | ca. 0-100 | |
| Malizia | |||||||
| Bleggiana | Trento | Bleggio Inferiore, Bleggio Superiore | ca. 9.6 | 1083 mm | 46°01’N;10°50’E | ca. 495-600 | |
| Feltrina | Belluno | Feltre | ca. 13 | 1520 mm | 46°01’N;11°53’E | ca. 300-500 | |
Analisi genetiche
Sia l’estrazione del DNA genomico sia l’amplificazione di marcatori ISSR sono state eseguite secondo i protocolli opportunamente messi a punto per il noce ([32]). Considerando i livelli relativamente bassi di polimorfismo precedentemente osservati in popolazioni e provenienze di noce ([14]), in questo studio sono stati usati undici primers (Tab. 2) invece dei nove già selezionati per la loro efficienza nel differenziare le quattro "varietà-modello" italiane ([32]). Pertanto, anche 20 campioni di ogni "varietà modello" già analizzati, sono stati amplificati con tutti gli undici primers; in totale, l’analisi genetica è stata condotta su 356 campioni. La Tab. 1 riassume le caratteristiche geografiche e climatiche dei 10 siti e delle 4 zone d’origine delle “varietà-modello”.
Tab. 2 - Lista degli 11 ISSR primers usati per la caratterizzazione genetica di 376 genotipi: Campania e Abruzzo e quattro varietà-modello italiane (Bleggiana, Feltrina, Sorrento, Malizia). Y = (C, T) V = (A, C, G) H = (A, C, T).
| Primer ISSR | Sequenza 5’- 3’ | N° bande | % polimorfismo | Range (bp) |
|---|---|---|---|---|
| UBC-807 | AGAGAGAGAGAGAGAGT | 14 | 57.1 | 246-1168.5 |
| UBC-810 | GAGAGAGAGAGAGAGAT | 12 | 58.3 | 467.4-1476 |
| UBC-811 | GAGAGAGAGAGAGAGAC | 12 | 75 | 360-1629.8 |
| UBC-834 | AGAGAGAGAGAGAGAGYT | 13 | 92.3 | 430.9-1599 |
| UBC-836 | AGAGAGAGAGAGAGAGYA | 13 | 61.5 | 381.3-1660 |
| UBC-841 | GAGAGAGAGAGAGAGAYC | 14 | 71.4 | 246-1537.5 |
| UBC-856 | ACACACACACACACACYA | 12 | 75 | 351.4-1968 |
| UBC-888 | BDBCACACACACACACA | 9 | 77.7 | 430.5-1199.25 |
| UBC-889 | DBDACACACACACACAC | 12 | 58.3 | 430.5-1230 |
| UBC-890 | VHVGTGTGTGTGTGTGT | 14 | 85.7 | 338.86-1476 |
| UBC-891 | HVHTGTGTGTGTGTGT | 9 | 100 | 405.9-1291.5 |
| Totale | - | 134 | 73.8 | - |
Analisi morfologica
Su un sub-set di 150 frutti, di cui 21 appartenenti alla varietà Bleggiana sono stati misurati alcuni tra i più comuni caratteri morfologici UPOV: A) Diametro equatoriale B) Diametro polare C) Rapporto tra A e B. Inoltre é stato valutato il peso secco del gheriglio.
Analisi biochimica
Le indagini sono state condotte sugli stessi frutti misurati per i caratteri morfologici.
- Estrazione Olio Totale: Il gheriglio di noci mature è stato disidratato mediante liofilizzazione e macinato in un mortaio di porcellana. L’olio totale è stato estratto con esano (1.5 ml/gr di tessuto). Dopo centrifugazione (7000 rpm, 20 min), il surnatante è stato raccolto ed il sedimento sottoposto ad estrazione per altre 3 volte. Dopo l’evaporazione dell’esano in corrente di azoto fino ad ottenere volume e peso costanti, è stata ottenuta la frazione pura di olio. Venti μl di olio sono stati risospesi in 1 ml di NaOH [1%] in metanolo ed idrolizzati a 80°C per 60 min. Dopo evaporazione sotto vuoto, il residuo è stato sciolto in 2 ml di acido solforico 0.15 N. Gli acidi grassi, solubilizzati in 1 ml di esano, sono stati analizzati in HPLC.
- Estrazione Proteine Totali: Le proteine totali sono state estratte dalle farine residue con NaOH 0.1M e quantificate con il metodo di Lowry usando siero-albumina-bovina come proteina standard.
- Analisi acidi grassi: L’identificazione e la quantificazione é stata fatta in HPLC (pompa Jasco Trirotar VI, rilevatore Jasco UV-975). I cromatogrammi alla lunghezza d’onda di 210 nm sono stati elaborati con un sistema Borwin. Le analisi effettuate su colonna Merck Purospher RP-18e (250x4 mm) eluita ad 1 ml/min con acetonitrile:isopropanolo:acqua (50:30:20) per 20 min e successivamente con acetonitrile:isopropanolo:acqua (50:45:5) per 10 min per verificare l’idrolisi completa dei trigliceridi.
Analisi dei dati
La matrice dei marcatori ISSR è stata costruita mediante il codice binario presenza (1), assenza (0) di una banda amplificata. Il confronto tra genotipi campionati e le 4 varietà modello é stato effettuato mediante il coefficiente di similarità Simple Match (SM - [37]) sulla cui matrice è stata condotta l’analisi delle coordinate principali (PcoordA). La distanza genetica tra le accessioni è stata quantificata mediante l’indice Population PhiPT value (F PT- [31]) e visualizzata mediante l’analisi PcoordA. La percentuale di varianza genetica spiegata tra ed entro accessioni é stata calcolata con l’analisi AMOVA ([10]), mentre per i caratteri morfologici e biochimici dei frutti, la ripartizione della varianza tra ed entro le accessioni è stata quantificata mediante l’analisi multivariata MANOVA. La correlazione statistica tra i dati morfologici, biochimici e genetici è stata valutata con il Test di Mantel ([27]) e il coefficiente di correlazione lineare di Pearson (r) è stato usato per la relazione tra le variabili morfologiche e biochimiche. Tutte le analisi sono state eseguite mediante i programmi GenAlEx V5.1 ([31]) e NTSYSpc version 2.1 ([36]).
Risultati
Marcatori ISSR
Gli undici primer ISSR utilizzati hanno amplificato in totale 134 frammenti. La Tab. 2 mostra il numero di frammenti amplificati per primer, il range in paia di basi e la percentuale di polimorfismo riscontrata.
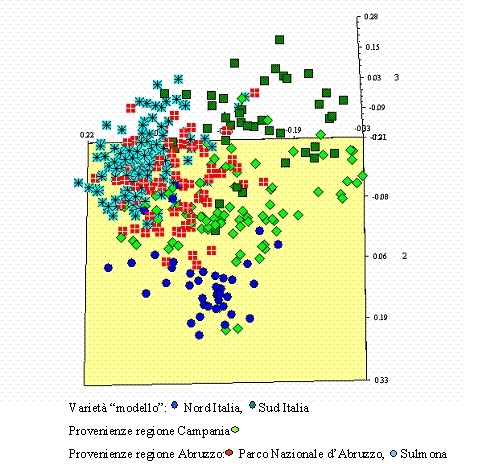
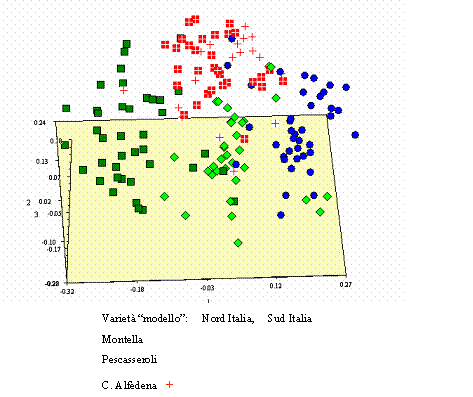
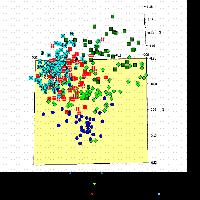
L’analisi delle cooordinate principali (PcoordA) condotta sulla matrice SM (Fig. 2) ha messo in evidenza le relazioni tra tutti i 356 individui analizzati. Le varietà del nord Italia (Bleggiana e Feltrina) sono separate da quelle del sud (Sorrento e Malizia) dalla prima e seconda coordinata principale. La maggior parte dei genotipi provenienti dalla Campania (Trescine Mesanole e Montella) sono riuniti al centro del plot, anche se alcune piante sono disperse tra le varietà modello e tra alcune accessioni abruzzesi. Il cluster più ampio include le piante campionate in Abruzzo (accessioni di Sulmona e del Parco Nazionale d’Abruzzo).
Fig. 2 - Analisi delle coordinate principali di 358 genotipi totali basata sulla matrice di similarità Simple Match calcolata su 134 ISSR.
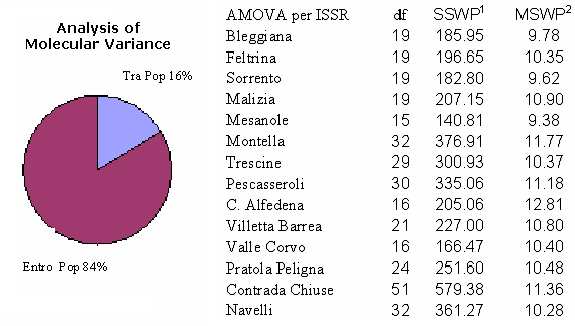
Nella Fig. 3 sono riportati i valori della varianza riscontrata tra ed entro le provenienze analizzate e le varietà modello. L’analisi AMOVA ha mostrato che la varianza è maggiore all’interno (83%) che tra (16%) le provenienze; il più alto livello di variabilità interna (MSWP2 = 12.81) è proprio di C. Alfedena, mentre i valori più bassi sono osservati per le varietà Bleggiana, Sorrento e l’accessione campana Mesanole (MSWP2 = 9.78, 9.62, 9.38, rispettivamente).
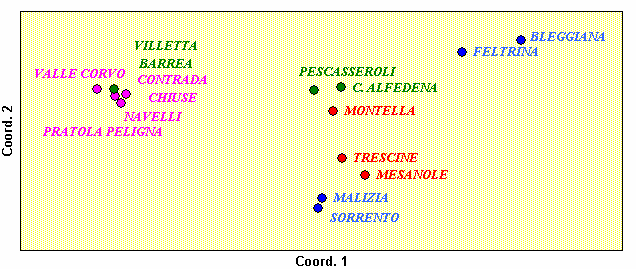
Con lo scopo di chiarire le relazioni esistenti tra le provenienze, abbiamo stimato la differenziazione genetica (DG) tra gruppi mediante l’indice F PT sui 134 ISSR totali (Tab. 3). Il valore DG più basso è quello tra Valle Corvo e Villetta Barrea (0.012), mentre il più elevato è tra le varietà Malizia e Bleggiana (0.322). L’analisi delle coordinate principali condotta su tale matrice (Fig. 4) ha mostrato, l’attesa e netta separazione delle “varietà modello” del nord da quelle del sud Italia secondo la prima e seconda coordinata principale. Riunite in uno stesso gruppo sono tutte le provenienze del comune di Sulmona e Villetta Barrea (Parco Nazionale d’Abruzzo); in posizione intermedia rispetto alle varietà e al gruppo precedente sono Pescasseroli e Civitella Alfedena (DG = 0.053) nelle cui vicinanze si colloca, sorprendentemente, la provenienza campana Montella (DG da Pescasseroli = 0.131; DG da Civ. Alfedena = 0.123). Le altre due accessioni della Campania, Trescine e Mesanole (DG = 0.089), sono più vicine al gruppo delle varietà modello del sud Italia.
Tab. 3 - Differenziazione genetica delle provenienze in base al valore F pt calcolato su 134 marcatori ISSR (livello di significatività 0.01% per 1000 permutazioni). Sopra la diagonale: livello di significatività F pt per ogni confronto tra provenienze
| Località | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | 14 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 - Bleggiana | 0.000 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 |
| 2 - Sorrento | 0.319 | 0.000 | 0.002 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 |
| 3 - Malizia | 0.322 | 0.058 | 0.000 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 |
| 4 - Feltrina | 0.128 | 0.282 | 0.251 | 0.000 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 |
| 5 - Mesanole | 0.289 | 0.196 | 0.207 | 0.265 | 0.000 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 |
| 6 - Montella | 0.184 | 0.201 | 0.180 | 0.140 | 0.116 | 0.000 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 |
| 7 - Trescine | 0.226 | 0.140 | 0.147 | 0.209 | 0.089 | 0.063 | 0.000 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 |
| 8 - Pescasseroli | 0.205 | 0.203 | 0.181 | 0.172 | 0.230 | 0.131 | 0.156 | 0.000 | 0.002 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 |
| 9 - C.Alfedena | 0.178 | 0.194 | 0.160 | 0.136 | 0.222 | 0.123 | 0.161 | 0.053 | 0.000 | 0.001 | 0.001 | 0.001 | 0.001 | 0.001 |
| 10 - V.Barrea | 0.279 | 0.213 | 0.195 | 0.230 | 0.222 | 0.111 | 0.148 | 0.133 | 0.142 | 0.000 | 0.158 | 0.001 | 0.001 | 0.001 |
| 11 - V.Corvo | 0.274 | 0.203 | 0.197 | 0.226 | 0.232 | 0.138 | 0.152 | 0.126 | 0.121 | 0.012 | 0.000 | 0.001 | 0.001 | 0.001 |
| 12 - P.Peligna | 0.298 | 0.229 | 0.229 | 0.244 | 0.249 | 0.162 | 0.193 | 0.163 | 0.154 | 0.096 | 0.080 | 0.000 | 0.001 | 0.001 |
| 13 - C.Chiuse | 0.257 | 0.213 | 0.205 | 0.209 | 0.206 | 0.111 | 0.148 | 0.147 | 0.146 | 0.040 | 0.060 | 0.042 | 0.000 | 0.009 |
| 14 - Navelli | 0.276 | 0.192 | 0.189 | 0.214 | 0.205 | 0.124 | 0.154 | 0.159 | 0.164 | 0.061 | 0.092 | 0.056 | 0.013 | 0.000 |
Fig. 4 - Analisi delle coordinate principali di 10 accessioni provenienti dalla regione Campania (Trescine, Mesanole, Montella) e Abruzzo (Valle Corvo, Contrada Chiuse, Pratola Peligna, Navelli, Villetta Barrea, C. Alfedena, Pescasseroli) e quattro varietà-modello (Bleggiana, Feltrina, Sorrento, Malizia) basata su F PT, usando 134 ISSR totali.
Osservando la Fig. 4 si nota una particolare similarità genetica tra l’accessione campana Montella e le provenienze abruzzesi Pescasseroli e C. Alfedena. Escludendo la possibilità di flusso genico e scambi di germoplasma tra le due regioni soprattutto per la relativa lontananza e le barriere geografiche, abbiamo approfondito l’analisi di dette accessioni includendo, sempre come riferimento, le 4 varietà modello. L’analisi delle coordinate principali condotta sulla matrice di similarità SM (Fig. 5) mostra che tra le varietà del nord e del sud Italia, separate tra loro dalla prima coordinata principale, si collocano due gruppi differenziati in base alla seconda e terza coordinata: un gruppo include le provenienze abruzzesi, l’altro riunisce la maggior parte delle piante di Montella anche se alcuni individui si mescolano con le varietà del nord e con l’Abruzzo.
Fig. 5 - Confronto tra le provenienze Pescasseroli, C. Alfedena e Montella e le quattro varietà-modello mediante l’Analisi delle Componenti Principali, basata su 134 ISSR.
Caratteri morfologici
Come già precisato, per tali dati preliminari come unica varietà di riferimento è stata arbitrariamente considerata solo Bleggiana. I valori medi dei caratteri morfologici dei frutti sono riportati in Tab. 4. I valori minimi di peso secco sono stati riscontrati in Pescasseroli (6.72 g ± 0.88) e Bleggiana (6.87 ± 1.43) mentre il valore massimo è in Navelli (10.27 g ± 1.14) Il rapporto diametro equatoriale/polare, da cui dipende la forma del frutto, suddivide il germoplasma in tre distinti gruppi: le accessioni di Sulmona e Villetta Barrea presentano frutti ovati-oblunghi; Pescasseroli e C. Alfedena sono caratterizzati da frutti tondeggianti come la varietà modello Bleggiana; Montella, Mesanole e Trescine producono frutti ellittici-oblunghi. L’analisi MANOVA ha evidenziato che il 12.6% della varianza è dovuta alle differenze tra accessioni, mentre l’87.4% è imputabile alla diversità individuale delle piante entro ogni accessione (dati non mostrati). Per verificare l’eventuale esistenza di una correlazione statistica tra i dati genetici e morfologici abbiamo condotto il test di Mantel e calcolato l’indice di correlazione di Pearson (r), la cui significativitàè stata stabilita in seguito a mille permutazioni casuali dei dati delle matrici. Esiste una correlazione debolmente significativa tra i dati genetici e morfologici (r = 0.369 e P = 0.021) con un livello di probabilità di 0.01% (Tab. 5).
Tab. 4 - Musure morfologiche e dati biochimici rilevati nei frutti delle accessioni analizzate e nella varietà-modello Bleggiana.
| (a) | |||||
| Morfologia | Diametro Equatoriale (A- mm) |
Diametro Polare (B - mm) |
Forma (A/B) |
Peso Secco (gr) |
|
|---|---|---|---|---|---|
| Pratola Peligna | 27.8 ± 1.59 | 31.53 ± 2.89 | 0.8816 | 7.63 ± 1.11 | |
| Valle Corvo | 30.31 ± 1.58 | 32.97 ± 2.01 | 0.9193 | 9.83 ± 1.3 | |
| Contrada Chiuse | 29.1 ± 2.06 | 31.75 ± 2.37 | 0.9165 | 9.00 ± 1.8 | |
| Navelli | 30.61 ± 2.92 | 33.43 ± 2.17 | 0.9156 | 10.11 ± 1.14 | |
| Villetta Barrea | 28.76 ± 2.73 | 30.72 ± 2.28 | 0.9361 | 7.97 ± 1.34 | |
| C.Alfedena | 28.23 ± 0.99 | 32.77 ± 2.41 | 0.8614 | 8.92 ± 0.74 | |
| Pescasseroli | 26.84 ± 1.3 | 31.02 ± 2.17 | 0.8652 | 6.72 ± 0.88 | |
| Bleggiana | 26.89 ± 2.44 | 30.92 ± 2.71 | 0.8696 | 6.87 ± 1.43 | |
| Montella | 30.61 ± 2.19 | 37.21 ± 2.89 | 0.8226 | 9.51 ± 1.75 | |
| Trescine | 29.18 ± 1.6 | 35.62 ± 3.23 | 0.8192 | 9.39 ± 1.46 | |
| Mesanole | 28.6 ± 1.41 | 36.62 ± 2.16 | 0.7809 | 9.62 ± 1.06 | |
| (b) | |||||
| Biochimica | Proteine Totali (mg/gr peso secco) |
Olio Totale (mg/gr peso secco) |
Acido ω-3 % |
Acido ω- 6 % |
Acido Oleico % |
| Pratola Peligna | 187.566 ± 36.68 | 529.32 ± 43.50 | 15.93 ± 2.26 | 60.56 ± 5.15 | 23.50 ± 4.36 |
| Valle Corvo | 178.897 ± 26.28 | 497.74 ± 74.80 | 16.62 ± 2.62 | 64.36 ± 3.95 | 18.92 ± 5.30 |
| Contrada Chiuse | 178.023 ± 36.47 | 520.17 ± 36.18 | 15.01 ± 4.06 | 63.09 ± 3.07 | 22.01 ± 1.66 |
| Navelli | 166.444 ± 29.31 | 497.00 ± 32.74 | 16.40 ± 2.93 | 61.80 ± 3.32 | 21.79 ± 4.37 |
| Villetta Barrea | 211.648 ± 32.03 | 517.69 ± 39.52 | 17.96 ± 3.98 | 59.46 ± 3.44 | 22.50 ± 3.54 |
| C.Alfedena | 225.569 ± 23.12 | 501.74 ± 43.20 | 16.97 ± 0.73 | 62.91 ± 2.25 | 20.16 ± 2.10 |
| Pescasseroli | 249.403 ± 31.79 | 513.93 ± 31.99 | 17.96 ± 2.93 | 60.12 ± 3.27 | 21.90 ± 3.75 |
| Bleggiana | 188.928 ± 37.12 | 525.70 ± 43.53 | 15.62 ± 2.20 | 57.42 ± 5.69 | 27.05 ± 5.88 |
| Montella | 237.110 ± 46.86 | 488.89 ± 50.32 | 16.52 ± 3.17 | 69.14 ± 4.45 | 14.35 ± 5.37 |
| Trescine | 226.617 ± 29.75 | 584.93 ± 40.80 | 19.96 ± 4.48 | 64.89 ± 2.81 | 15.18 ± 4.28 |
| Mesanole | 244.326 ± 70.91 | 528.82 ± 49.42 | 17.97 ± 2.05 | 66.26 ± 5.40 | 15.71 ± 5.19 |
Tab. 5 - (a) Coefficiente di Pearson per lo studio della correlazione tra i caratteri quantitativi analizzati; (b) Correlazione statistica tra dati qualitativi (marcatori ISSR) e quantitativi secondo il test di Mantel (1000 permutazioni). (*): p<0.05; (**): p<0.01; (ns): non significativo.
| (a) | |||||||||
| Coefficiente r | Diametro Equatoriale | Diametro Polare | Peso Secco | Forma | Proteine Totali | Olio Totale | Acido ω- 3 | Acido ω- 6 | Acido Oleico |
|---|---|---|---|---|---|---|---|---|---|
| D. Equatoriale | 1 | - | - | - | - | - | - | - | - |
| D. Polare | 0.566ns | 1 | - | - | - | - | - | - | - |
| Peso Secco | 0.888** | 0.701* | 1 | - | - | - | - | - | - |
| Forma | 0.128ns | -0.743** | -0.125ns | 1 | - | - | - | - | - |
| Proteine Totali | -0.277ns | 0.428ns | -0.153ns | -0.731* | 1 | - | - | - | - |
| Olio Totale | -0.305ns | 0.097ns | -0.130ns | -0.359ns | 0.175ns | 1 | - | - | - |
| Acido ω- 3 | -0.009ns | 0.370ns | 0.118ns | -0.445ns | 0.628b* | 0.598ns | 1 | - | - |
| Acido ω- 6 | 0.660* | 0.921** | 0.743** | -0.572ns | 0.382ns | -0.087ns | 0.231ns | 1 | - |
| Acido Oleico | -0.567ns | -0.912** | -0.682* | 0.634* | -0.548ns | -0.133ns | -0.557ns | -0.936** | 1 |
| (b) | |||||||||
| Mantel Test | r - Product-moment correlation | P level | |||||||
| Genetica x Morfologia | 0.3690 | 0.021* | |||||||
| Genetica x Biochimica | 0.170 | 0.185ns | |||||||
| Morfologia x Biochimica | 0.358 | 0.016* | |||||||
Caratteri biochimici
Sui frutti collezionati nella stagione 2003-2004 è stata effettuata un’analisi preliminare biochimica dei principali composti: proteine totali, olio totale, acidi grassi polinsaturi (Tab. 4). Come già precisato, per tale analisi come varietà di riferimento è stata arbitrariamente considerata la Bleggiana. Il contenuto percentuale di olio rispetto al peso secco del frutto è risultato tra 48.8% di Montella e 58.5% di Trescine, mentre le proteine totali sono comprese tra il valore massimo di Pescasseroli (249 mg g-1 DW ± 31.7) ed il minimo di Navelli (166 ± 29.31 mg g-1 DW). Una decisa variabilità si riscontra nella composizione percentuale degli acidi grassi polinsaturi che in forma esterificata compongono gli oli dei frutti. La media di acido oleico e linoleico (w-6) sull’olio totale oscilla rispettivamente tra 14.3 - 27.0% e 57.8 - 69.1%. I frutti di Montella contengono più w-6 (69.14 ± 4.45) e meno acido oleico (14.35 ± 5.37) rispetto a quelli delle altre accessioni; al contrario le noci della “varietà-modello” Bleggiana hanno il maggiore contenuto di acido oleico (27.05 ± 5.88) e il livello più basso di w-6 (57.82 ± 5.69). I valori di deviazione standard assai elevati suggeriscono una considerevole diversità biochimica tra i genotipi delle varie accessioni. La MANOVA sui dati biochimici infatti stabilisce che il 99.94% della varianza è dovuta a differenze tra individui mentre appena il 0.06% alla diversità tra gruppi. Non esiste correlazione statistica tra i dati biochimici e quelli genetici (r = 0.170, P = 0.185 ns), mentre risulta significativa ma di modesto rilievo la correlazione tra i dati biochimici e morfologici (r = 0.358, P = 0.016).
Discussione
Due accessioni della Campania, Trescine e Mesanole, presentano un’elevata similarità genetica, morfologica e biochimica con le due varietà-modello, Sorrento e Malizia, originarie entrambe della Penisola Sorrentina. La varietà Sorrento è famosa per la qualità del frutto di forma ellittica-oblunga (diametro equatoriale A: 32 ± 2 mm, polare B: 41.7 ± 2.47 mm, rapporto A/B: 0.75-0.78), elevato peso secco (11 ± 1 g), olio 55-60% DW ([5]; [25]), con una percentuale di w-6, w-3 e di acido oleico rispettivamente del 68%, 17% e del 15%. Questi valori sono congruenti con quelli ottenuti in Trescine e Mesanole. Poiché condividono la stessa area di distribuzione con le varietà-modello del Sud Italia, è lecito supporre che le due accessioni, composte da genotipi nati da seme locale, siano strettamente correlate alla varietà Sorrento.
Le piante localizzate nel circondario di Sulmona a differenti altitudini (Navelli 685 m, Contrada Chiuse 500 m, Valle Corvo 430 m, Pratola Peligna 330 m) ed a Villetta Barrea (Parco Nazionale d’Abruzzo, 1000 m) formano un unico cluster, e si dividono dal resto della collezione sulla base dei marcatori ISSR. Questo risultato trova conferma anche nella forma ovata - oblunga dei frutti appartenenti alle stesse popolazioni. Tuttavia, a causa della diversità tra gli individui, non risulta alcuna struttura genetica interna al gruppo. La differenziazione in base ai caratteri molecolari e morfologici delle accessioni di Sulmona potrebbe essere il risultato della selezione di genotipi locali, destinati alla produzione di frutti o di legno di qualità, mentre si può supporre che l’azione diretta dell’ambiente sia irrilevante. Inoltre, l’accessione Villetta Barrea, pur localizzandosi tra quelle del Parco Nazionale d’Abruzzo, è composta da piante probabilmente provenienti dalla vicina Valle Corvo. Lo scambio di materiale vegetale può essere avvenuto in passato grazie alla "transumanza” che si praticava in quelle zone. I pastori, che in estate trasferivano le loro greggi dalla valle di Sulmona ai pascoli del Parco Nazionale d’Abruzzo, tra vettovaglie ad alto valore nutrizionale possono aver trasportato, e quindi diffuso vicino agli stalli, anche le noci (Cannata F, comunicazione personale).
Montella, si distingue dalle altre provenienze campane mentre risulta geneticamente più simile a Pescasseroli e C. Alfedena, due località del Parco Nazionale d’Abruzzo. Poiché Campania e Abruzzo sono, come già detto, due regioni separate dagli Appennini, sembra improbabile che vi sia stato flusso di polline e/o semi tra tali siti geografici. La similarità perciò potrebbe essere il risultato di un adattamento degli individui a condizioni microclimatiche simili. Le piante raccolte a C. Alfedena che invece si mescolano con Pescasseroli potrebbero derivare da un insieme di semi di origine incerta, trasportati dall’uomo in quell’area; a ciò potrebbe essere dovuto la variabilità interna piuttosto elevata riscontrata per tale accessione. Pescasseroli e Montella che geneticamente si differenziano dalle altre provenienze, presentano anche particolari combinazioni di caratteri morfo-biochimici del frutto. La noci di Montella sono di forma ellittica - oblunga, dal gheriglio carnoso, con un più alto rapporto w-6 / acido oleico, come le varietà-modello Sorrento e Maliza; ma hanno una bassa concentrazione di olio totale. Interessante risulta la percentuale di w-6 che supera i valori di riferimento presenti in letteratura per la specie J. regia ([40], [43], [6]). Tanto nei semi quanto nelle membrane plastidiali è stata riscontrata una relazione inversa tra il grado di insaturazione degli acidi grassi contenuti e la loro temperatura di crescita ([20], [29], [35]). La proporzione di w-6 nel seme aumenta al diminuire della temperatura di sviluppo (chilling acclimatation), in relazione anche all’area geografica e alla stagione ([8]). Questa relazione osservata nei semi di girasole ([19], [24]), Chartamus tinctorius ([23]), soia ([21]), rapa ([39]), Borrago officinalis ([18]) è dovuta alla dipendenza dalla temperatura dell’enzima w-6 desaturasi, che, dall’acido oleico, catalizza la sintesi di w-6. Montella, che si trova in una valle appenninica (670 m), con temperature relativamente basse (media annua 12.8°C) ed abbondanti precipitazioni (1341.3 mm), potrebbe essere un ecotipo naturalmente adattato a tali condizioni climatiche.
Le piante collezionate a Pescasseroli presentano frutti di pezzatura medio-piccola, con livelli relativamente alti di acido oleico e bassi di w-6 (come la varietà Bleggiana), ma un elevato contenuto di proteine totali che supera i valori riportati in precedenti lavori ([41], [6]). In numerose specie vegetali quali la mela ([7]), il melone ([22]), mais, pisello e girasole ([17]), è stato osservato che il tasso di espansione di un frutto è direttamente proporzionale alla temperatura a cui avviene lo sviluppo. Pescasseroli si trova nel Parco Nazionale d’Abruzzo a 1200 m di altitudine, con una temperatura media di 7.8°C e abbondanti precipitazioni annue (1520 mm). Questi dati potrebbero indicare un adattamento di tali piante a basse temperature, anche se il livello di w-6 non è alto. A tal proposito però, non bisogna trascurare il ruolo svolto dagli antiossidanti a e g tocoferolo nell’impedire la conversione di w-6 in acido oleico. Sembra infatti che la quantità di tocoferolo nelle accessioni abruzzesi sia decisamente più bassa (dati non mostrati).
La correlazione, se pur di bassa entità, tra marcatori molecolari e caratteri morfologici, come anche quella tra morfologia e biochimica, è particolarmente interessante. Mentre sembra, infatti, confermata l’eredità materna per la forma e la grandezza del frutto, non appare nessun legame tra la madre e il contenuto di acidi grassi e proteine. Tali composti, che sono sostanze di riserva del seme, potrebbero essere sotto il controllo di più geni che si attivano allorquando occorrono particolari condizioni pedoclimatiche. Tuttavia tali deduzioni sono da considerarsi soltanto a livello di supposizioni poiché i marcatori genetici ISSR utilizzati, essendo neutrali, non necessariamente descrivono la diversità genetica di natura adattativa. Per approfondire lo studio delle correlazioni tra caratteri qualitativi e quantitativi sarebbe quindi necessario ripetere l’analisi con marcatori funzionali e, disponendo di opportuno materiale, con l’analisi di geni candidati.
Tuttavia, in base ai risultati ottenuti, potremmo formulare l’ipotesi che Montella e Pescasseroli siano realmente due ecotipi su cui approfondire le indagini e che esse potrebbero essere valorizzate come fonte di acidi grassi essenziali, e come accessioni da seme adatte per la riforestazione di zone montane e alta collina.
Ringraziamenti
La ricerca è stata svolta nell’ambito del Programma di ricerca: Agro-Biotecnologie, Progetto: Valorizzazione della biodiversità frutticola mediterranea - BIOFRUM, Legge 449/97, 1999 del Ministero Università e Ricerca (MIUR).
References
Online | Google Scholar
Google Scholar
CrossRef | Google Scholar
Online | Google Scholar
Google Scholar
Google Scholar
Google Scholar
Google Scholar
Online | Google Scholar
Online | Google Scholar
Online | Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
Google Scholar
Google Scholar
Google Scholar
Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
Online | Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
Google Scholar
Google Scholar
Online | Google Scholar
Online | Google Scholar
Google Scholar
CrossRef | Google Scholar
Online | Google Scholar
Google Scholar
Google Scholar
Google Scholar
CrossRef | Google Scholar
Google Scholar
Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
Google Scholar
Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar