Global change and upward shift of treeline in the Alps: genetic consequences of pastures colonization
Forest@ - Journal of Silviculture and Forest Ecology, Volume 4, Pages 69-78 (2007)
doi: https://doi.org/10.3832/efor0431-0040069
Published: Mar 21, 2007 - Copyright © 2007 SISEF
Research Articles
Guest Editors: 5° SISEF Congress (Grugliasco, TO - 2005)
« Forests and Society - Changes, Conflicts, Sinergies »
Collection/Special Issue: E. Lingua, R. Marzano, G. Minotta, R. Motta, A. Nosenzo, G. Bovio
Abstract
In the Alps global warming and change in land use, in particular the drastic reduction in cattle grazing, are causing a progressive treeline ecotone upwards shift. The colonization dynamics of woody species are modulated by high selective pressure on seedlings due to the extreme ecological conditions of this habitat. We studied the colonization dynamics of Norway spruce in a treeline plot, at the upper limit of the Paneveggio forest (Trentino, Italy). We have exhaustively sampled the study stand, collecting needle tissue from all the adults and the juveniles detected, and we have genotyped all the samples (376) with 4 SSR markers. Parentage relationships between the few adults presents in the stand (23) and the juveniles were established. Our results indicate that, in spite of extreme ecological condition of the treeline environment, local adaptations do not seem to favour local parents: only 4% of juveniles have both parents among local adult trees, while 96% of juveniles are completely or partially sired outside the sampling area. Assessing parentage relationship also allowed the estimation of relative reproductive success of local adult trees. Out of a total of 23 adult trees, 5 trees were involved in the 59% of successful reproductive events detected inside the sampling area.
Keywords
Introduzione
Negli ultimi decenni stiamo assistendo ad un progressivo innalzamento delle temperature medie ([28], [68]), e si prevede che questo trend aumenterà rapidamente nei prossimi anni ([69]). Le condizioni climatiche influenzano l’ecofisiologia, la produttività e la distribuzione delle specie arboree ([67]). Gli ambienti d’alta montagna, come le Alpi, sono particolarmente vulnerabili ai cambiamenti climatici ([65]). Le temperature minime annue nelle Alpi sono aumentate di 2 °C nell’ultimo secolo, soprattutto dagli anni Ottanta in poi ([3]). Inoltre, nelle aree alpine stiamo assistendo ad un progressivo abbandono dei pascoli d’altura ([15]). L’effetto congiunto di questi due fattori sta promuovendo l’innalzamento dell’ecotono di treeline, la zona tra il limite superiore continuo del bosco e gli ultimi individui isolati ritrovabili a quote superiori ([34]).
Il processo di colonizzazione nelle piante forestali è stato studiato con un approccio modellistico da Austerlitz et al. ([2]) e i risultati del modello sviluppato hanno mostrato che, per specie caratterizzate da uno stadio giovanile prolungato e dal conseguente ritardo del primo evento riproduttivo, le prima fase della colonizzazione di una nuova area è supportata principalmente dall’arrivo di nuovi migranti dalle popolazioni sorgente, piuttosto che dalla riproduzione degli individui colonizzatori insediati. Questa dinamica determina una scarsa differenziazione genetica tra la popolazione sorgente e quella neofondata, tendendo a diminuire l’effetto del fondatore. I risultati del modello trovano riscontro in recenti lavori riguardanti specie arboree ([42], [54], [11], [18], [36]).
Vi sono però pochi studi riguardanti la variabilità genetica in popolazioni di specie forestali disposte lungo transetti altitudinali, tutti eseguiti con marcatori enzimatici, che hanno evidenziato sia l’esistenza di elevata variabilità tra quote differenti per determinati loci ([25], [40]), sia una decrescita della variabilità all’aumentare della quota ([64], [52]). La spiegazione maggiormente plausibile di tali risultati è che la selezione imponga sia una perdita di variabilità al crescere della quota, dovuta alle difficoltà di insediamento di genotipi provenienti da quote inferiori, che causerebbe un marcato effetto “collo di bottiglia” (bottleneck); sia un gradiente di varianti genotipiche adattate alle differenti condizioni ecologiche e microambientali presenti lungo transetti altitudinali ([47]). In tale scenario l’effetto della selezione contrasterebbe le conseguenze dell’elevato flusso genico caratteristico delle specie forestali, specialmente in quelle ad impollinazione e disseminazione anemofila ([26]).
L’ambiente di treeline riveste grande importanza principalmente per l’alta pressione selettiva sperimentata dalle specie residenti, dovuta alle condizioni ecologiche estreme che lo contraddistinguono (basse temperature, elevata ventosità, copertura nevosa prolungata - [16]), e per l’elevata sensibilità nei confronti delle variazioni di temperatura ([67]). Nonostante ciò non sono mai stati eseguiti lavori riguardanti la genetica della colonizzazione legata allo shift altitudinale delle foreste.
La genetica della colonizzazione può venire studiata finemente analizzando i pattern del flusso genico e la loro influenza sulla struttura genetica nell’area di espansione. Esistono due approcci principali, basati sull’utilizzo di marcatori molecolari, allo studio della microstrutturazione spaziale: lo studio della distribuzione spaziale di alleli e/o genotipi ([17]) e la stima diretta del flusso genico mediante l’analisi di paternità o di parentela ([31]). Recentemente, gli studi sulle relazioni di parentela sono stati notevolmente incentivati dallo sviluppo di marcatori molecolari altamente polimorfici, come i microsatelliti (SSR), e di nuove procedure statistiche ([43], [22], rivisti da [31]). Nelle specie forestali i marcatori genetici sono stati utilizzati soprattutto per stimare la distanza di dispersione del polline e l’efficienza d’impollinazione mediante l’analisi di paternità svolta direttamente sui semi campionati sulle piante madri ([14], [63], [21], [55]). Ma Dow & Ashley ([13]) hanno sottolineato, nonostante l’importanza dello studio delle dinamiche d’impollinazione, come la fitness degli organismi studiati dipenda dalla prole insediata con successo e che sopravvive nei primi stadi di crescita. Di conseguenza, la determinazione delle relazioni di parentela negli individui già insediati rappresenta l’unico approccio che permette di evidenziare gli eventi di dispersione geneticamente ed ecologicamente rilevanti.
In questo studio abbiamo eseguito l’analisi di parentela in un’area di treeline nella foresta di abete rosso nel Parco Naturale di Paneveggio-Pale di San Martino, utilizzando un approccio probabilistico “most likely”. Questa metodica è stata sviluppata per assegnare la progenie analizzata ai potenziali genitori non escludibili in base ad incompatibilità genetica, attribuendo la parentela in modo categorico al genitore o alla coppia di genitori più probabile ([45]). I risultati permettono di determinare quali siano i pattern di impollinazione e di disseminazione nell’area di studio, il flusso genico proveniente dall’esterno, il successo riproduttivo e l’eventuale esistenza di relazioni tra successo riproduttivo e variabili morfo-fisiologiche nelle piante adulte presenti nell’area.
Materiali e Metodi
Specie studiata
L’abete rosso (Picea abies (L.) Karst.) è una delle specie forestali più comuni ed economicamente importanti d’Europa. Il suo areale naturale si estende dalla Francia ad ovest (longitudine 5° E) fino agli Urali (55° E) ad est, e dalla Norvegia (latitudine 70° N) a nord fino alla Macedonia (42° N) a sud ([57]). L’areale può venire suddiviso in due macroaree principali: un’area boreale nord-est europea e un’area montana centroeuropea ([24]). La struttura genetica di P. abies rispecchia questa suddivisione ([66]; [61]; [12]), supportando l’ipotesi di ricolonizzazione da due rifugi glaciali formulata in base ai dati palinologici ([29]). In Italia l’abete rosso si ritrova su tutto l’arco alpino e in alcune piccole popolazioni nell’Appennino settentrionale ([4]).
La maggior parte degli studi molecolari su questa specie hanno riguardato la distribuzione della variabilità genetica in macroaree ([1]); al contrario, sono state scarsamente studiati la microstrutturazione spaziale ed i pattern del flusso genico contemporaneo (ad eccezione di [37], [8], [9]).
Area di studio
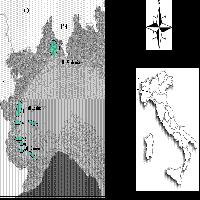
L’area di studio si trova nella foresta di Paneveggio (latitudine 46°18’ N, longitudine 11°45’ E), sulle pendici del monte Cavallazza (2324 m s.l.m.) nella parte superiore della Valbona. La temperatura media annua è 2.4 °C e le precipitazioni annuali 1316 mm a Passo Rolle ([20]), a 3 km dall’area di studio. La copertura nevosa persiste a 1700 m s.l.m. per circa 5 mesi ([10]). La foresta è stata utilizzata intensamente per l’alpeggio fino agli anni Settanta, successivamente il pascolo è stato abbandonato progressivamente e l’area è stata ripopolata dal camoscio, dal capriolo e dal cervo ([49]); la crescente densità delle popolazioni di ungulati selvatici sta determinando ingenti danni alla rinnovazione all’interno della foresta ([48]).
L’area di studio è un plot di 16 ha (200 m x 800 m) che si estende altitudinalmente dal limite continuo del bosco (Laghi del Colbricon, 1927 m s.l.m.) sulle pendici del Monte Cavallazza fino all’ultimo individuo di abete rosso che siamo stati in grado di individuare al limite superiore della zona di colonizzazione (2200 m s.l.m.). Nell’area è stato riscontrato un gradiente di morfotipi tipico delle zone di treeline: da individui di piccole dimensioni a portamento arbustivo (krummholz) nella fascia superiore (Fig. 1), fino alle morfologie classiche di grandi dimensioni e a portamento arboreo al limite continuo del bosco.
Strategia di campionamento
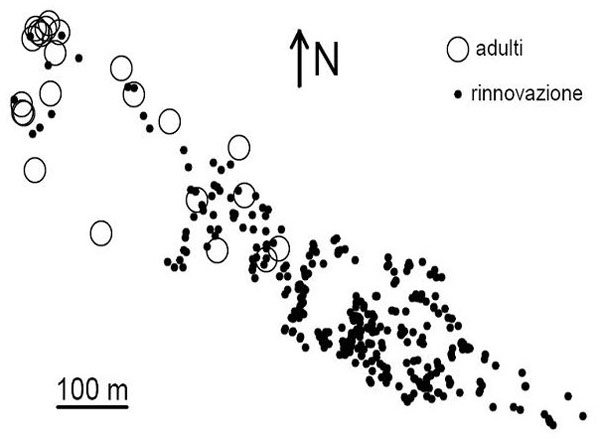
In totale sono stati prelevati campioni di tessuto (aghi) da 376 abeti, rappresentanti tutti gli individui presenti nella zona di colonizzazione e tutti gli individui adulti della fascia altitudinale immediatamente sottostante. Gli individui campionati sono stati classificati come potenziali genitori (23) e rinnovazione (353) in base al fatto che sia stata riscontrata la presenza di strobili sulla chioma. Tutti gli individui campionati sono stati mappati utilizzando un distanziometro laser Leica DISTO3 ed una bussola (Fig. 2). Inoltre, di ogni individuo campionato sono state misurate altezza e circonferenza alla base, utilizzando un ipsometro laser Haglöf Vertex III e una cordella metrica.
Fig. 2 - Mappa degli individui campionati. Gli adulti (cerchi grandi vuoti) si trovano nella fascia altitudinale inferiore del plot, mentre la rinnovazione (cerchi piccoli pieni) si estende fino a circa 2200 m s.l.m.
Analisi genetica
Da 70 mg di ogni campione di aghi è stato estratto il DNA, utilizzando il kit di estrazione Qiagen Plant DNeasy96. La genotipizzazione è avvenuta utilizzando quattro marcatori microsatellite (SSR): tre sviluppati da Scotti et al. ([59]) e uno da Pfeiffer et al. ([51]). La PCR (polymerase chain reaction) è stata effettuata utilizzando un thermal cycler PTC100 (MJ Research) in un volume totale di 25 µl. Per i primer EATC1B2, EATC2B2 e EATC1E3 è stata utilizzando una mix di reazione contenente 10 ng di DNA, 100 μM di ciascun dNTP, 10X PCR buffer (100mM Tris-HCl (pH 8.8 a 25°C), 500mM KCl, 0.8% Nonidet P40), 2.5 mM di MgCl2, 0.1 μM di primer forward e 0.25 μM di primer reverse e 1 U di Taq Polimerasi (Fermentas). Per SpAGG03 è stata utilizzando una mix di reazione contenente 10 ng di DNA, 100 μM di ciascun dNTP, 10X PCR buffer (100mM Tris-HCl (pH 8.8 a 25°C), 500mM KCl, 0.8% Nonidet P40), 2.5 mM di MgCl2, 0.2 μM di ciascun primer e 1 U di Taq Polimerasi (Fermentas). Le condizioni di reazione per la PCR sono state: 95 °C per 10 min, 7 cicli “touch-down” (95 °C per 30 sec, Ta + 7 °C per 30 sec, 72 °C per 30 sec), 28 cicli di amplificazione (95 °C per 45 sec, Ta per 45 sec, 72 °C per 45 sec) e uno step finale di estensione di 72 °C per i primer EATC1B2, EATC2B2 e EATC1E3. Le condizioni di reazione per la PCR per SpAGG03 sono state: 94 °C per 10 min, 34 cicli di amplificazione (94 °C per 45 sec, Ta per 45 sec, 72 °C per 45 sec) e uno step finale di estensione di 10 min a 72 °C. I prodotti di PCR sono stati analizzati mediante elettroforesi capillare sul sequenziatore CEQ2000 della Beckman Coulter.
Analisi dei dati
Per ogni locus sono state calcolate le frequenze alleliche, l’eterozigosi attesa (He) in base alla formula proposte da Hartl & Clark ([27]), il numero di alleli (Na), il numero di alleli privati (Npa, [41]), e il numero effettivo di alleli (Ne = 1 / (1 - He), [7]).
La differenziazione genetica tra potenziali genitori e rinnovazione è stata misurata utilizzando i coefficienti FST e RST ([60]) calcolati mediante l’AMOVA (Analysis of Molecular Variance - [19]). L’RST è un analogo dell’FST stimato in base allo stepwise mutation model, un modello specifico per marcatori microsatellite che tiene in considerazione le differenze di dimensione tra gli alleli ([46]). La significatività dei risultati è stata valutata mediante 1000 permutazioni, seguendo il metodo di Lindenmayer & Peakall ([39]). Per tutte le analisi genetiche è stato utilizzato il software GenAlEx v. 6 ([50]).
L’analisi di parentela è stata svolta utilizzando il software FaMoz ([23]). In primo luogo è stata calcolata la probabilità di esclusione del nostro set di marcatori, seguendo la formula proposta da Jamieson & Taylor ([30]). Successivamente per tutte le possibili combinazione singolo genitore/figlio e coppia di genitori/figlio è stato calcolato il LOD (likelihood ratio) score, cioè il rapporto tra la probabilità di un singolo adulto/coppia di adulti di essere il genitore/genitori di un determinato individuo giovane, e la probabilità che tale relazione sia casuale ([22]).
Con il software utilizzato è possibile determinare la soglia oltre la quale i LOD score vengono considerati statisticamente significativi. Tale soglia viene calcolata simulando una progenie proveniente dall’esterno rispetto all’area campionata e confrontando la distribuzione dei conseguenti LOD score con quelli ottenuti sui dati reali. La soglia è rappresentata dall’intersezione delle curve delle due distribuzioni ([22]). Successivamente, utilizzando un “categorical allocation approach” ([31]), la parentela è stata assegnata alle coppie o ai singoli genitori aventi il LOD score statisticamente significativo maggiore.
Assegnata la parentela è stato possibile quantificare sia la dispersione all’interno dell’area campionata, sia il flusso genico dall’esterno. In particolare, il tasso di immigrazione dall’esterno è stato calcolato sia come numero di individui originatisi interamente o parzialmente fuori dall’area di studio, sia a livello gametico come il numero di gameti prodotti all’esterno sul numero totale di gameti campionati.
Inoltre, grazie ai risultati dell’analisi di parentela è stato possibile calcolare le distanze di disseminazione e di dispersione del polline all’interno dell’area di studio, basandosi sulle assunzioni presentate da Dow & Ashley ([13]): (i) se un individuo giovane è compatibile con un solo genitore, allora questo genitore viene considerato il produttore del seme; e (ii) se un individuo giovane è compatibile con una coppia di genitori, il genitore più vicino viene considerato il produttore del seme, mentre quello più lontano il produttore del polline.
Infine, è stato calcolato il successo riproduttivo di ogni potenziale genitore all’interno dell’area di studio considerando il numero totale di eventi riproduttivi (via polline o via seme) attribuiti ad un individuo. La relazione tra il successo riproduttivo e i tratti morfologici misurati (altezza e diametro alla base) è stata indagata utilizzando un modello lineare generalizzato basato sulla distribuzione di Poisson ([44]). La significatività del modello è stata testata utilizzando l’analisi sequenziale della devianza.
L’elaborazione statistica dei dati risultanti dall’analisi di parentela è stata effettuata utilizzando il pacchetto statistico R ([53]).
Risultati
I 4 marcatori microsatellite sono risultati essere altamente variabili, con un numero medio di 12.5 alleli per locus sull’intero set di dati. La frazione di individui giovani (rinnovazione) è caratterizzata da una quota rilevante di alleli che non sono presenti nei genitori (7.5 in media), mentre nessun allele privato è stato riscontrato tra i potenziali genitori (Tab. 1). La differenziazione genetica tra potenziali genitori e rinnovazione è risultata essere molto bassa, con un valore di FST di 0.009 e di RST di 0.03.
Tab. 1 - Variabilità genetica nei potenziali genitori e nella rinnovazione locus per locus (Na = numero di alleli; Ne = numero effettivo di alleli; Npa = numero di alleli privati; He = eterozigosi attesa).
| Stage | Locus | Na | Ne | Npa | He |
|---|---|---|---|---|---|
| Potenziali genitori | SpAGG03 | 12 | 7.389 | 0 | 0.865 |
| EATC1B2 | 4 | 1.418 | 0 | 0.295 | |
| EATC1E3 | 5 | 2.396 | 0 | 0.583 | |
| EATC2B2 | 9 | 3.796 | 0 | 0.737 | |
| Rinnovazione | SpAGG03 | 17 | 9.463 | 5 | 0.894 |
| EATC1B2 | 12 | 1.694 | 8 | 0.41 | |
| EATC1E3 | 11 | 2.512 | 6 | 0.602 | |
| EATC2B2 | 20 | 2.788 | 11 | 0.641 |
Le probabilità di esclusione del nostro set di marcatori (0.994 per le coppie di genitori e 0.815 per i singoli genitori) sono sufficientemente alte per discriminare tra l’esiguo numero di individui adulti presenti nell’area.
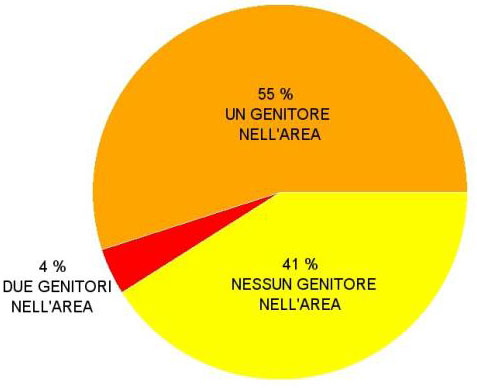
Analizzando i LOD score che sono risultati essere maggiori rispetto alla soglia di significatività calcolata mediante 10000 simulazioni, si è evidenziato che tra i 353 individui giovani presenti nell’area di studio 210 individui (59%) sono compatibili con almeno un potenziale genitore presente nell’area di studio. Tra di essi 196 (55%) sono compatibili con un solo genitore, mentre 14 (4%) sono compatibili con una coppia di genitori. Ben 143 individui giovani (41%) sono invece risultati provenire interamente dall’esterno (Fig. 3). A livello gametico ciò significa che il 68% dei gameti campionati sono frutto di flusso genico dall’esterno.
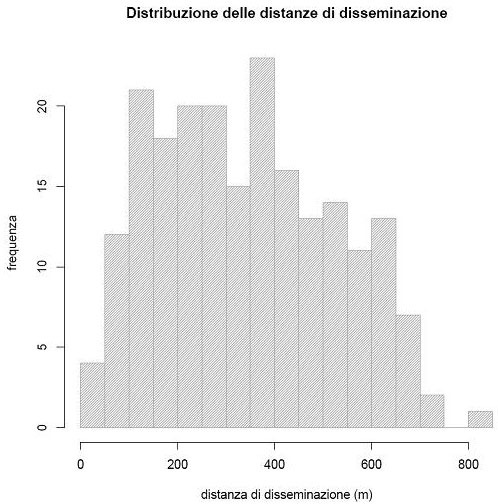
In base alle assunzioni di Dow & Ashley ([13]), la distanza media di disseminazione nell’area è risultata essere 341.95 ± 184.15 m (Fig. 4).
Fig. 4 - Distribuzione delle distanze di disseminazione relative agli eventi riproduttivi evidenziati con l’analisi di parentela nell’area di studio.
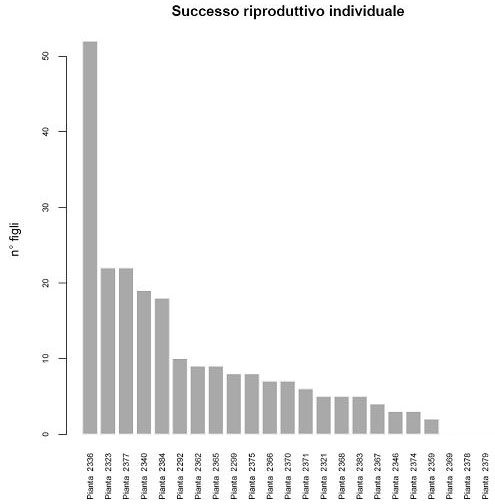
Il successo riproduttivo medio dei potenziali genitori nell’area è di 9.74 figli, e varia da 0 (le piante 2369, 2378 e 2379 non risultano essere genitori di alcun individuo giovane) fino ai 52 figli della pianta 2336, che risulta essere di gran lunga l’individuo col maggior successo riproduttivo (Fig. 5). Il successo riproduttivo è risultato essere dipendente dal diametro delle piante (c 2 [1]=12.114, P<0.001) ma non dall’altezza (c 2 [1]=0.875, P=0.349). Il diametro, valutato nel modello a parità di altezza, ha un effetto positivo sul successo riproduttivo (b = 0.0191 ± 0.0052 SE, P<0.001). Questo implica che apparentemente alla treeline, a parità di altezza, piante con fusto più largo abbiano un vantaggio su individui più affusolati.
Fig. 5 - Variabilità nel successo riproduttivo tra gli individui adulti presenti nell’area di studio.
Discussione
In questo studio vengono analizzati i pattern del flusso genico in un’area di treeline che sta venendo colonizzata dall’abete rosso. La causa di questo spostamento è probabilmente legata sia all’abbandono del pascolo d’altura che allo shift altitudinale favorito dal riscaldamento globale.
I nostri risultati dimostrano chiaramente che nella zona di treeline il flusso genico provenente dall’esterno è molto elevato. Infatti, sebbene il 59% della rinnovazione abbia almeno un potenziale genitore compatibile nel plot, solamente per il 4% degli individui giovani si è riusciti a non escludere almeno una coppia di genitori all’interno dell’area di studio, e, soprattutto, nel 96% dei casi almeno metà del corredo genetico proviene da individui collocati all’esterno. Questi dati confermano che, nonostante l’elevata pressione selettiva tipica dell’ecotono di treeline ([16]), un’alta percentuale dei geni che partecipano alla colonizzazione proviene dall’esterno, molto probabilmente da individui della foresta sottostante, confermando l’ipotesi che la sopravvivenza degli individui giovani non provenienti dalla treeline non sia inferiore rispetto a quella dei figli delle piante già insediate a livello della treeline. Uno scenario simile può venire interpretato in base all’enorme potenziale dispersivo dell’abete rosso, i cui propaguli (polline e seme) vengono entrambi trasportati dal vento. Molti autori hanno dimostrato come in particolare il polline delle conifere possa viaggiare per decine o centinaia di chilometri ([38], [56]). La conseguenza genetica di questa caratteristica è che le piante anemofile sono caratterizzate da una scarsissima differenziazione genetica tra popolazioni e da una grande variabilità entro popolazione ([33], [1]). In uno studio di paternità su un popolazione piccola e isolata di Pinus sylvestris, specie con impollinazione anemofila, è stato dimostrato che, nonostante la popolazione più vicina fosse a 30 km, il 5% del flusso pollinico proveniva dall’esterno rispetto all’area di studio ([55]); mentre nell’abete rosso, con un approccio simile all’analisi di parentela, Burczyk et al. ([9]) hanno dimostrato che all’interno di una seed orchard di 0.89 ha l’83% dei semi è stato impollinato da polline proveniente dall’esterno.
Un’altra possibile spiegazione al fatto che individui provenenti dalla foresta sottostante siano in grado di insediarsi in un’area caratterizzata da condizioni assai differenti da quelle in cui sono stati prodotti, potrebbe risiedere nel fatto che le condizioni attuali di temperatura alla treeline siano più favorevoli per genotipi provenienti dalla fascia altitudinale immediatamente sottostante il limite continuo del bosco. Infatti, come sottolineato da Billington & Pelham ([5]) e Jump & Peñuelas ([32]), in generale il flusso genico da aree più calde potrà rivelarsi fondamentale per l’adattamento alle nuove condizioni ambientali determinate dall’attuale innalzamento delle temperature medie.
L’elevato flusso genico e la differenziazione riscontrata tra adulti presenti alla treeline e rinnovazione (FST = 0.009 e RST = 0.03), molto bassa sebbene più elevata rispetto a quella normalmente riscontrata per l’abete rosso tra popolazioni geograficamente anche molto distanti ([1]), confermano le attese del modello di Austerlitz et al. ([2]). Infatti, i nostri risultati indicano che l’avanzamento del fronte di colonizzazione viene principalmente supportato dal flusso genico dalla popolazione sorgente, e non dai pochi individui adulti insediati alla treeline.
Per quanto riguarda il successo riproduttivo, è interessante notare che la distribuzione del numero di figli tra le piante adulte risulta fortemente sbilanciato, con la pianta 2336 compatibile come genitore con 52 individui giovani, e quindi coinvolta ben nel 23% degli eventi riproduttivi riscontrati nell’area di studio. Questa percentuale sale al 59% se si considerano le 5 piante adulte con maggiore successo riproduttivo. Un risultato simile è stato riscontrato da Dow & Ashley ([13]) in Quercus macrocarpa, una specie però caratterizzata da una limitata capacità di disseminazione, testimoniata dal fatto che gli individui giovani siano stati tutti localizzati immediatamente attorno alla pianta d’origine. Il successo riproduttivo degli adulti alla treeline è risultato essere significativamente correlato al diametro delle piante. In studi precedenti basati su dati genetici sono stati ottenuti risultati contrastanti riguardanti la relazione tra successo riproduttivo e dimensioni delle piante. In Dicorynia guianensis, una specie tropicale, è stata evidenziata una relazione positiva tra il successo riproduttivo e il diametro ([35]). Il volume della chioma è invece risultato essere solo debolmente associato al successo riproduttivo in Quercus macrocarpa ([14]). In Gleditsia triachantos, la fertilità femminile è risultata dipendente dalle dimensioni della pianta solo in una delle due aree contigue studiate da Schnabel et al. ([58]). Alla treeline potrebbe essere più conveniente investire in crescita radiale che in altezza per sfruttare maggiormente la protezione della copertura nevosa e le condizioni favorevoli che essa determina a livello del suolo, evitando i danni causati dai forti venti, dalle tempeste di ghiaccio e dalla siccità invernale ([62]), oppure le piante di diametro maggiore potrebbero semplicemente avere avuto il vantaggio di essere stati i primi colonizzatori, producendo così una progenie più numerosa semplicemente poiché coinvolti in un maggior numero di eventi riproduttivi. Però si deve tener conto che alla treeline le dimensioni della pianta non ne rispecchiano necessariamente l’età, a causa dei vincoli che le particolari condizioni ambientali impongono alla crescita, e gli individui più prolifici potrebbero essere stati solamente favoriti, ad esempio, dalla loro posizione nell’area studiata. Per meglio comprendere le cause del pattern del successo riproduttivo riscontrato sarà interessante stimare l’età delle piante attraverso analisi dendrocronologiche, al fine di valutare se le differenze nel successo riproduttivo siano imputabili ad un diverso numero di stagioni riproduttive attraversate dalle piante adulte, oppure caratterizzare genotipicamente le piante adulte con marcatori mappati su sequenze espresse, i cui trascritti sono coinvolti in processi metabolici importanti in ambienti altamente selettivi, per raccogliere eventuali prove a favore dell’esistenza di adattamenti alle condizioni della treeline ([6]).
Infine, i risultati di questo studio, da considerarsi preliminari, potranno venire chiarificati sia aumentando il numero dei marcatori molecolari utilizzati e, di conseguenza, le probabilità di esclusione sulla base delle quali viene svolta l’analisi di parentela, sia ampliando il campionamento analizzando individui adulti nella foresta sottostante per valutare quale sia il contributo relativo delle fasce altitudinali inferiori all’insediamento della rinnovazione alla treeline.
Ringraziamenti
La ricerca è stata supportata dai progetto MURST-COFIN 2002: “Foreste di alta quota delle Alpi e degli Appennini: strutture, fattori limitanti, adattamenti fisiologici e dinamiche passate, presenti e future”, coordinato dal Prof. Renzo Motta. Gli autori ringraziano Patrizia Rossi, Beppe Storti, Roberta Berretti, il Prof. Renzo Motta e il personale della Stazione Forestale di Paneveggio (TN) per avere contribuito all’organizzazione e alla logistica dell’attività di campo, Marilena Meloni per le informazioni riguardanti i marcatori utilizzati e il Prof. Pietro Piussi per gli utili commenti su una prima versione del manoscritto.
References
CrossRef | Google Scholar
Google Scholar
CrossRef | Google Scholar
Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
Google Scholar
CrossRef | Google Scholar
Google Scholar
CrossRef | Google Scholar
Google Scholar
Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
Google Scholar
Google Scholar
Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
Google Scholar
Google Scholar
Google Scholar
Google Scholar
Google Scholar
CrossRef | Google Scholar
Google Scholar
Google Scholar
Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
Google Scholar
CrossRef | Google Scholar
Google Scholar
Google Scholar
Google Scholar
CrossRef | Google Scholar
Online | Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
Online | Google Scholar
Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
CrossRef | Google Scholar
Google Scholar